gwas-kinship
亲缘系数(coefficient of kinship):从两个个体中分别随机抽取一个同源等位基因,这两个基因是IBD的概率(相同且来自同一个祖先)。直接衡量个体间亲缘关系。
近交系数(inbreeding coefficient, F):一个个体某个基因座上的两个等位基因为血缘同源(IBD)的概率。衡量父母间亲缘关系。也称固定系数fixation index。
血缘系数(coefficient of relationship,r ):有共同祖先的两个个体基因型一致的概率。=2 fold coefficient of kinship
reference: https://gwaslab.com/2021/05/22/coefficient-of-kinship-inbreeding-relationship/
遗传分化指数Fst(Wahlund effect:通过Hardy-Weinberg来进行检验,如果很多个亚群被抽样,即便每一个亚群都符合Hardy-Weinberg平衡,那么抽样合并的总体也会偏离Hardy-Weinberg很远。而且这种偏离方向是一样的,即我们观测到的杂合子的数量总是比平衡预期的要少。这种杂合子欠缺的现象就是Wahlund effect(Wahlund 1928)。)
(选择信号)->群体结构
群体结构矫正:genomic control,PCA
亲缘关系矩阵(kinship)可以用于群体结构的可视化和关联分析中矫正群体结构混淆造成的高假阳性。我们通过计算个体i和个体j之间的亲缘关系系数来估计亲缘关系矩阵,这种方法是Amin 、Astle 和Balding Amin 所提出的(Amin et al., 2007 , Astle and Balding, 2009):
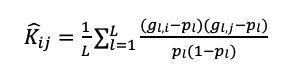
其中L代表marker的位置,是第l位的等位基因频率,是在第l位的第j个样本的基因型(取值0、1、2,分别代表小等位基因纯合、杂合和主等位基因纯合基因型)。
亲缘关系矩阵的计算是通过GenABLE包中的ibs函数实现的 (Aulchenko et al., 2007)。
加性效应:(AA-aa)/2,即育种值。
显性效应:Aa-(AA+aa)/2
为什么IBS可以矫正群体结构:
SNP连锁,corr=1,删除掉相同的部分,剩下的部分才能定到QTL。
为什么IBS计算后对角线是大概0.98?
SNP密度影响IBS?
单个染色体影响IBS?