gwas-model
Q(群体结构)+K(亲缘关系)模型
FASTA(why “permutation procedures can not be applied to estimate genome-wide significance, because the data structure is not exchangeable.”)
GRAMMAR(Amin et al, 2007)
EIGENSTRAT(GenABEL qtscore)
FASTA(GenABEL mmscore)
区分SNP、mutation、variant、allele、genotype。Allele frequency、Genotype frequency,alternative allele frequency、MAF。
我们使用混合线性模型对eQTL进行定位:
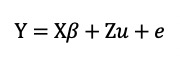
其中Y为中心化和标准化后的表达表型,属于正态分布,均值为0;X为nx2的固定效应的设计矩阵,其中n=298,第一列代表为单位向量,第二列代表每个生态型所关联SNP的等位基因计数(取值0、1、2,分别代表小等位基因纯合、杂合和主等位基因纯合基因型)的矩阵;beta为长度为2的向量,代表群体均值和SNP的加性效应;Z是随机效应的设计矩阵,通过对亲属矩阵G的Cholesky分解得到,其中G是使用GenABEL软件包中的ibs函数(option weight = ‘freq’)对全基因组MAF过滤过的SNP数据估计而来的(Aulchenko et al. ,2007 );e为随机残差。由于Z矩阵满足ZZ’=G,因此随机效应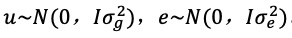 。
。
我们使用R语言中GenABEL包(Aulchenko et al., 2007)内置的polygenic 和mmscore(mixed model approximation analysis)函数进行以上的计算。
GRAMMARS
矩阵版模型:
plink、tassel使用的模型?
what are ‘lambda’,’P’,’PVE’,’effect size’?
基因组膨胀因子(inflation factor,lambda):用于GWAS结果的质控。基因组膨胀会因群体结构不充分而产生的,定义为所有SNP的卡方检验统计量的中值与卡方分布的预期中值之比。其统计原理为,当原假设成立时,每个SNP位点与表达性状都无关联,每个统计量都服从 1个自由度的卡方分布,所以所有SNP的卡方检验统计量的中值与从1个自由度的卡方分布中随机抽取的统计量中值之比应该为1。偏离1的eGWAS结构均会受到群体结构影响,因此我们在最终的结果中只保留lambda值为1±0.05的基因。
PVE estimated by REML
包含上位效应的模型?
SNP密度影响peak?会影响,因为LD的存在。理想情况下snp的密度为每个LD区间一个。
单个染色体peak?会影响kinship的计算
QQplot的意义:qqplot横坐标为uniform distribution的p值,纵坐标为obversed p。若为y=x的斜线,性状分布为uniform distribution,即只有遗传漂变,若不是,说明有基因选择,即我们想要寻找的位点。
P-value:In summary, a p-value is composed of three parts:
1) The probability random chance would result in the observation.
2) The probability of observing something else that is equally rare.
3) The probability of observing something rarer or more extreme.
R2=0.9:The relationship between the two variables explains 90% of the variation in the datal”